Charakterisierung von Proteinlösungen
Die genaue Charakterisierung von Proteinlösungen ist für eine optimale Prozessauslegung sowie Formulierungsentwicklung unabdingbar. Ein kritischer Parameter in der Entwicklung von Biopharmazeutika ist die Löslichkeit des Moleküls während des Prozesses und die Langzeitstabilität der pharmazeutischen Formulierung. Unsere Arbeitsgruppe beschäftigt sich deshalb intensiv mit der Untersuchung von Protein-Protein Wechselwirkungen, dem daraus resultierenden Phasenverhalten, der Vorhersagbarkeit des Phasenverhaltens und dessen Manipulation. Untersucht werden Modellproteine, pharmazeutisch relevante Moleküle wie beispielsweise Antikörper, unterschiedliche Enzymklassen, sowie Viren und virenähnliche Moleküle in ideal verdünnter Lösung sowie in hochkonzentrierten Formulierungen.
Literatur
Proteinphasenverhalten
Für die biopharmazeutische Industrie ist die Stabilität der biologisch aktiven Komponenten während der Verarbeitung, Formulierung und Lagerung von maßgeblicher Bedeutung, um die Patientensicherheit und Produktaktivität zu gewährleisten. Ein wichtiger Stabilitätsaspekt ist hierbei das Phasenverhalten des Zielmoleküls. Es kann zwischen dem löslichen, dem gelierten, dem kristallinen und dem ausgefällten Phasenzustand unterschieden werden. Kontrollierte Kristallisation und Fällung bieten der biopharmazeutischen Industrie eine kostengünstige Alternative zu herkömmlichen Trennverfahren und sind für Formulierungszwecke anerkannt. Unerwünschte Aggregation kann hingegen zu Produktverlusten und Veränderungen der dreidimensionalen Struktur des Zielmoleküls führen. Daher ist Wissen über das Phasenverhalten von Proteinen für die Downstream-Prozessentwicklung unabdingbar.
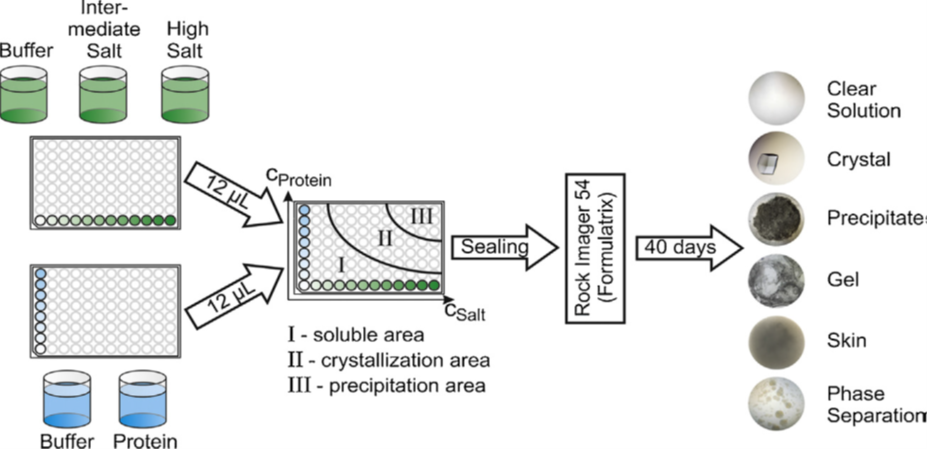
An unserem Institut wurde aus diesem Grund ein Hochdurchsatz-Verfahren entwickelt, was es erlaubt, Phasendiagramme auf einer automatisierten Liquid-Handling-Station im Mikroliter-Maßstab zu generieren. Unter Verwendung einer hochauflösenden Kamera werden die erzeugten Phasendiagramme über einen langen Zeitraum hinsichtlich ihres Phasenverhalten ausgewertet. Mit Hilfe dieser Methode kann der Einfluss verschiedener Prozessparameter, wie Art und Konzentration von Salzen oder Polymere, pH-Wert oder der Temperatur für verschiedene biologisch aktive Komponenten untersucht werden.